Cyanin5-Azid
| Artikel-Nr. | Packungseinheit | Preis | Vorlaufzeit | Jetzt kaufen |
|---|---|---|---|---|
| 13030 | 100 uL, 10 mM/DMSO |
–
|
Auf Lager | |
| 33030 | 500 uL, 10 mM/DMSO |
$199
|
Auf Lager | |
| 43030 | 1 mL, 10 mM/DMSO |
$495
|
Auf Lager | |
| A3030 | 1 mg |
–
|
Auf Lager | |
| B3030 | 5 mg |
$199
|
Auf Lager | |
| C3030 | 10 mg |
$297
|
Auf Lager | |
| D3030 | 25 mg |
$495
|
Auf Lager | |
| E3030 | 50 mg |
$895
|
Auf Lager | |
| F3030 | 100 mg |
$1490
|
Auf Lager |

Cyanin5-Azid (ein Analogon zu Cy5®-azid) ist ein Markierungsreagenz für die Click-Chemie, erhältlich als 10-mM-Lösung in DMSO und als Feststoff. Dieses Azid ist in organischen Lösungsmitteln (z. B. DMSO, DMF) löslich, das heißt die Markierungsreaktion ist in Gegenwart eines kleinen Anteils des organischen Lösungsmittels durchzuführen. Dieses Azid kann für die Markierung von Alkin-modifizierten Biomolekülen in Gemischen aus Wasser und organischen Lösungsmitteln verwendet werden. Die Lösung in DMSO ist gebrauchsfertig für die Anwendung in der Biokonjugation. Eine wasserlösliche Variante dieses Reagenzes ist ebenfalls erhältlich.
Cyanin5 ist einer der am häufigsten verwendeten Fluorophore und ist mit vielen Detektionsgeräten kompatibel. Cyanin5 ist eine Alternative zu Cy5® und DyLight 649.
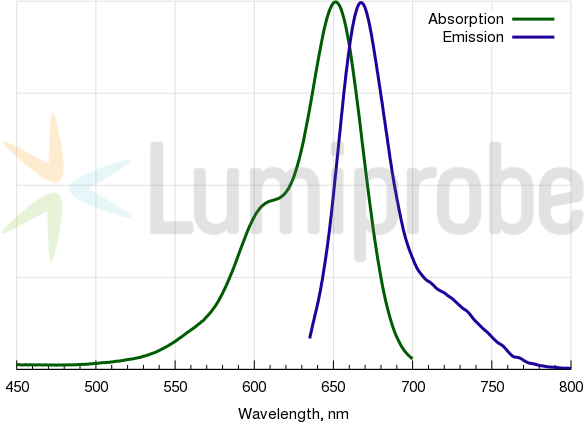
Absorptions- und Emissionsspektren von Cyanin 5
Kunden kauften zusammen mit diesem Produkt
Allgemeine Eigenschaften
| Erscheinungsform: | dunkelblaues Pulver bzw. Lösung |
| Molekülmasse: | 601.22 |
| CAS-Nummer: | 1267539-32-1 (chloride) |
| Molekülformel: | C35H45ClN6O |
| Löslichkeit: | löslich in organischen Lösungsmitteln (DMSO, DMF, Dichlormethan), geringe Löslichkeit in Wasser |
| Qualitätskontrolle: | NMR 1H, HPLC-MS (95 %) |
| Lagerungsbedingungen: | Lagerbeständigkeit: 24 Monate ab dem Wareneingang bei −20 °C an einem lichtgeschützten Ort. Transport: bei Raumtemperatur bis zu drei Wochen. Längere Lichteinwirkung vermeiden. Trocken lagern. |
| Sicherheitsdatenblatt: | herunterladen |
| Product specifications |
Spektrale Eigenschaften
| Anregungs-/Absorptionsmaximum / nm: | 646 |
|
ε |
250000 |
| Emissionsmaximum / nm: | 662 |
| Fluoreszenz-Quantenausbeute: | 0.2 |
| CF260: | 0.03 |
| CF280: | 0.04 |
Zitierungen
- Xu, M.J.; Jordan, P.W. SMC5/6 Promotes Replication Fork Stability via Negative Regulation of the COP9 Signalosome. International Journal of Molecular Sciences, 2024, 25(2), 952. doi: 10.3390/ijms25020952
- Ryl, T.; Afanasyeva, E.; Hartmann, T.; Schwermer, M.; Schneider, M.; Schröder, C.; Wagemanns, M.; Bister, A.; Kanber, D.; Steenpass, L.; Schramm, K.; Jones, B.; Jones, D.T.W.; Biewald, E.; Astrahantseff, K.; Hanenberg, H.; Rahmann, S.; Lohmann, D.R.; Schramm, A.; Ketteler, P. A MYCN-driven de-differentiation profile identifies a subgroup of aggressive retinoblastoma. Communications Biology, 2024, 7, 919. doi: 10.1038/s42003-024-06596-6
- Ramezani, M.; Bauman, J.; Singh, A.; Weisbart, E.; Yong, J.; Lozada, M.; Way, G.P.; Kavari, S.L.; Diaz, C.; Haghighi, M.; Batista, T.M.; Pérez-Schindler, J.; Claussnitzer, M.; Singh, S.; Cimini, B.A.; Blainey, P.C.; Carpenter, A.E.; Jan, C.H.; Neal, J.T. A genome-wide atlas of human cell morphology. bioRxiv, 2023. doi: 10.1101/2023.08.06.552164
- Tsuchiya, M.; Tachibana, N.; Hamachi, I. Flow cytometric analysis of phosphatidylcholine metabolism using organelle-selective click labeling. STAR Protocols, 2023, 4(3), 102525. doi: 10.1016/j.xpro.2023.102525

























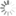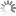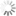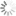
 $
$ 
